利用Python读取fasta文件并进行一系列操作(二)
概述:
本节目标:通过上一节所输出的txt输出ABO蛋白以及ABO基因的外显子fasta文件
语言:python3.8
模块:Biopython, ssl
可选:jupyter
整体思路:通过ncbi获取ABO基因外显子位置(爬虫),并读取txt,根据位置信息获取外显子序列,再通过外显子
序列输出mRNA序列以及蛋白序列
前排提示:本教程不管生物,有知识盲区自己补
步骤:
1. 设置ssl,要不debug后总会有bug
2. 从这步开始调用Biopython,设置邮箱(不要瞎填!!瞎填还不如不填!!!)
3. 设置有关搜索ncbi的函数以及参数,获取人类ABO基因的mRNA相关资料所对应id
4. 获取人类ABO基因的mRNA相关资料
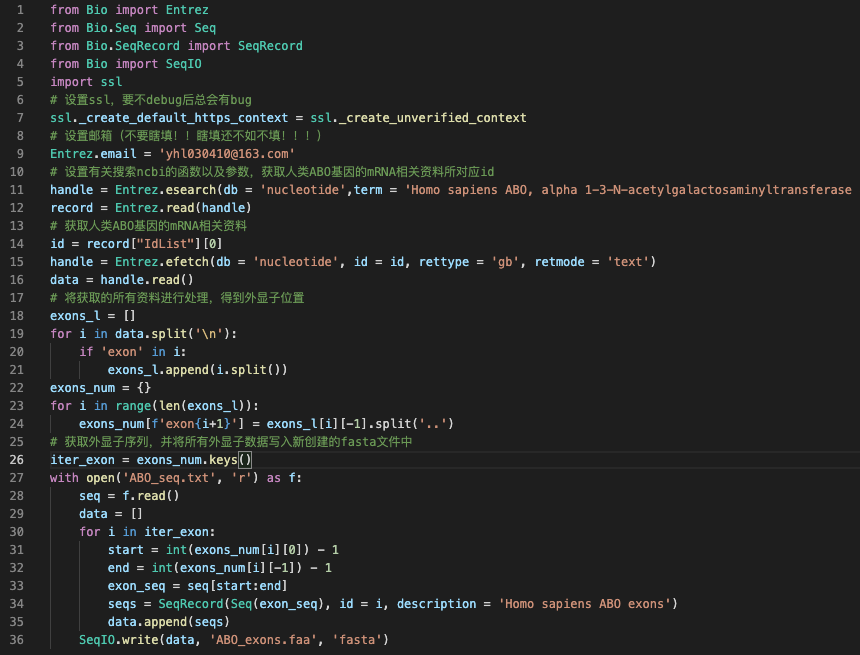
5. 将获取的所有资料进行处理,得到外显子位置
6. 获取外显子序列,并将所有外显子数据写入新创建的fasta文件中
代码:
结果展示:
获取蛋白序列继续往下看:
接上面步骤:
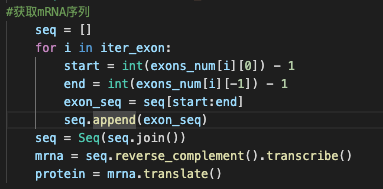
7. 将获取的外显子序列用Seq函数转为biopython类型
8. 将外显子倒序并转录
9. 将转录出的mRNA序列翻译为蛋白质序列
代码:
本节目标:通过上一节所输出的txt输出ABO蛋白以及ABO基因的外显子fasta文件
语言:python3.8
模块:Biopython, ssl
可选:jupyter
整体思路:通过ncbi获取ABO基因外显子位置(爬虫),并读取txt,根据位置信息获取外显子序列,再通过外显子
序列输出mRNA序列以及蛋白序列
前排提示:本教程不管生物,有知识盲区自己补
步骤:
1. 设置ssl,要不debug后总会有bug
2. 从这步开始调用Biopython,设置邮箱(不要瞎填!!瞎填还不如不填!!!)
3. 设置有关搜索ncbi的函数以及参数,获取人类ABO基因的mRNA相关资料所对应id
4. 获取人类ABO基因的mRNA相关资料
5. 将获取的所有资料进行处理,得到外显子位置
6. 获取外显子序列,并将所有外显子数据写入新创建的fasta文件中
代码:
结果展示:
获取蛋白序列继续往下看:
接上面步骤:
7. 将获取的外显子序列用Seq函数转为biopython类型
8. 将外显子倒序并转录
9. 将转录出的mRNA序列翻译为蛋白质序列
代码:



1 个回复
赞同来自:
要回复问题请先登录或注册