HGG.SNV数据库批量获取信息
HGG.SNV是一个了解不同人群中人类单核苷酸变异的进化和医学意义的数据库。这个数据库的亮点是它综合了很多项目的各种测序数据,其中包括HGDP、HapMap、gnomAD.genomes、SGDP等项目数据。其中包括220147份基因组数据,古人基因组1018份,覆盖了977个族群的268890619个位点。另一个亮点是有多种线上工具,操作方便。
网站链接:https://www.pggsnv.org/
但是当我们要查询的数据量十分庞大时会比较费时间,所以我们写一段python脚本来批量获取数据。
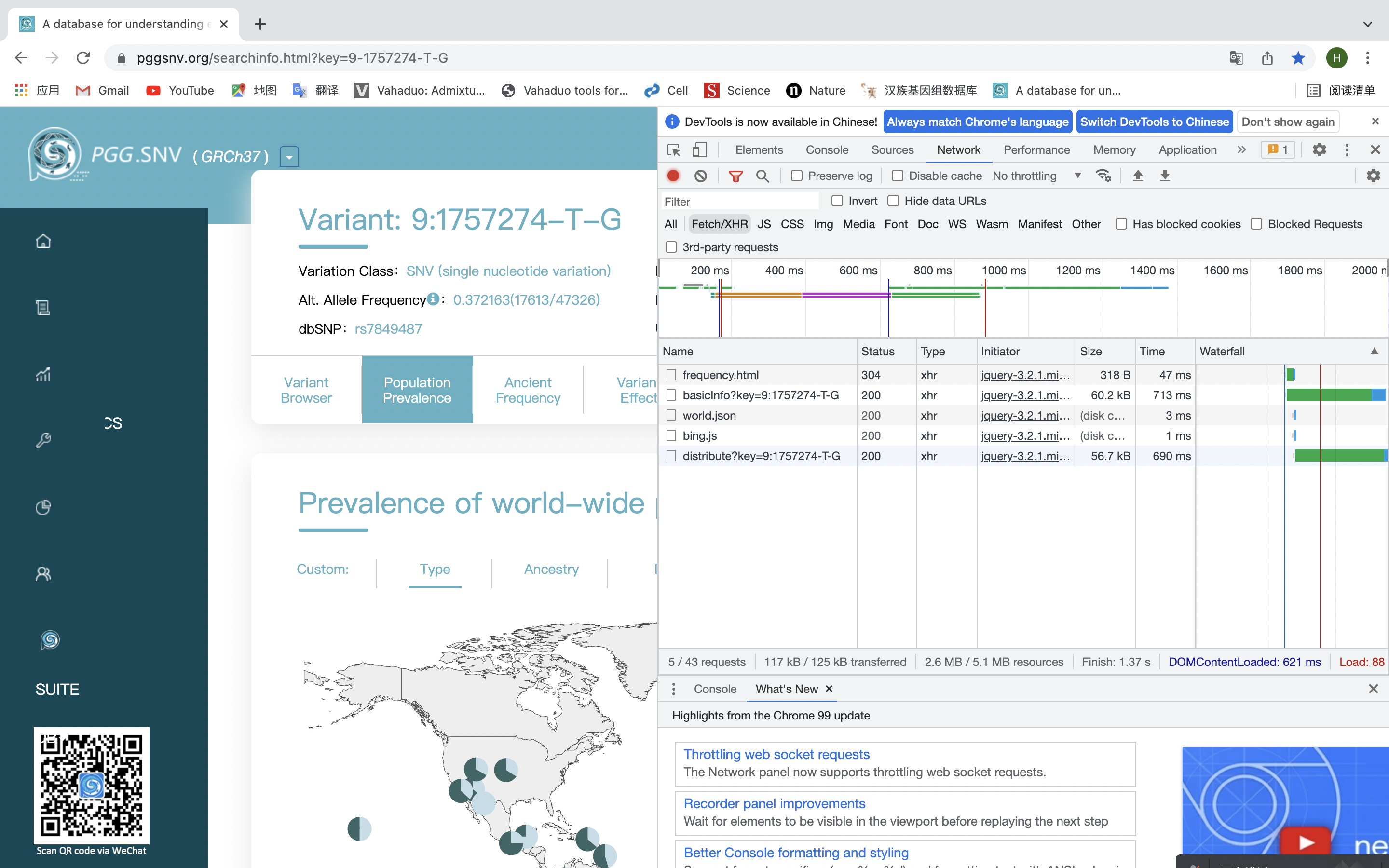
首先使用chrome对该网站的基本情况进行分析。使用Chrome的开发者工具对传输数据进行监控,并将传输数据筛选为fetch和XHR模式,发现该网站的前后端是通过json传输的,属于前后端分离的情况,所以我们可以直接向网站拉去请求获取json。
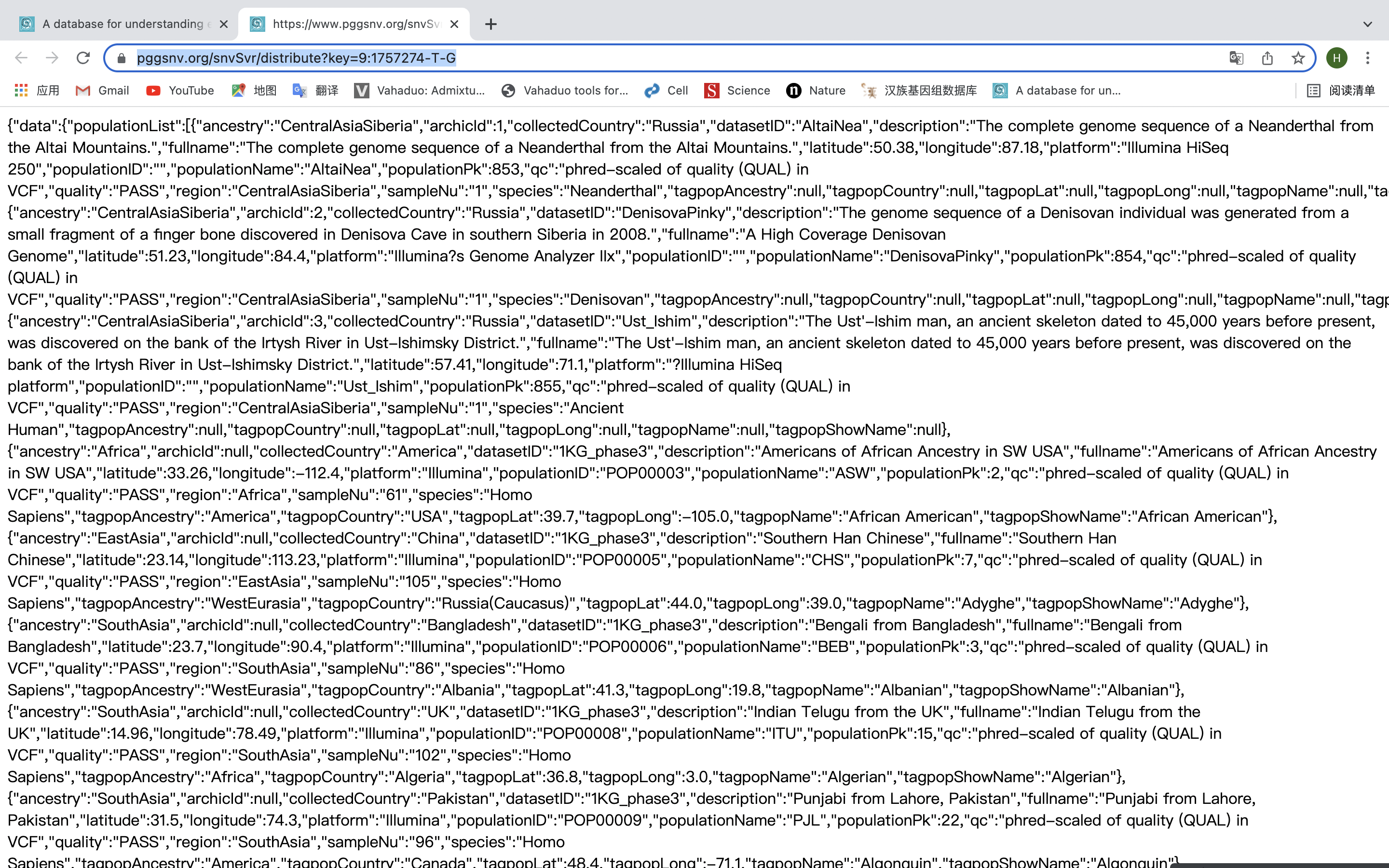
我们将其中的json打开,发现里面确实含有位点相关信息,所以双击该json文件获取url。
url:https://www.pggsnv.org/snvSvr/distribute?key=1:231557623-G-C
接着我们对url进行解析。通过对比已知位点数据可知该url中的key应该是以这样的格式组成的:染色体号:碱基对位置-ref-alt
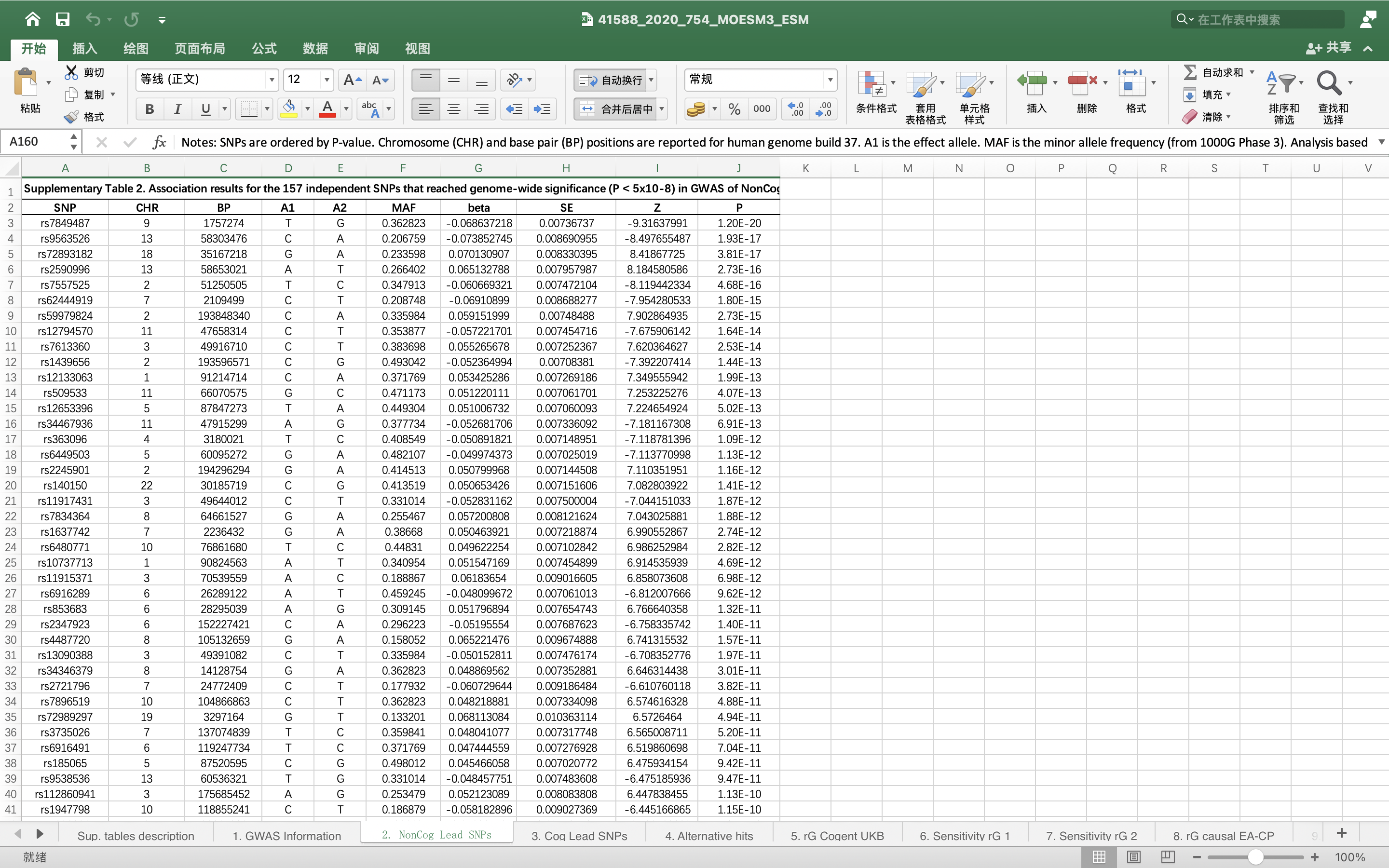
我们以《Nature-Genetics》期刊上的《Investigating the genetic architecture of noncognitive skills using GWAS-by-subtraction》【1】这篇文章的数据为例
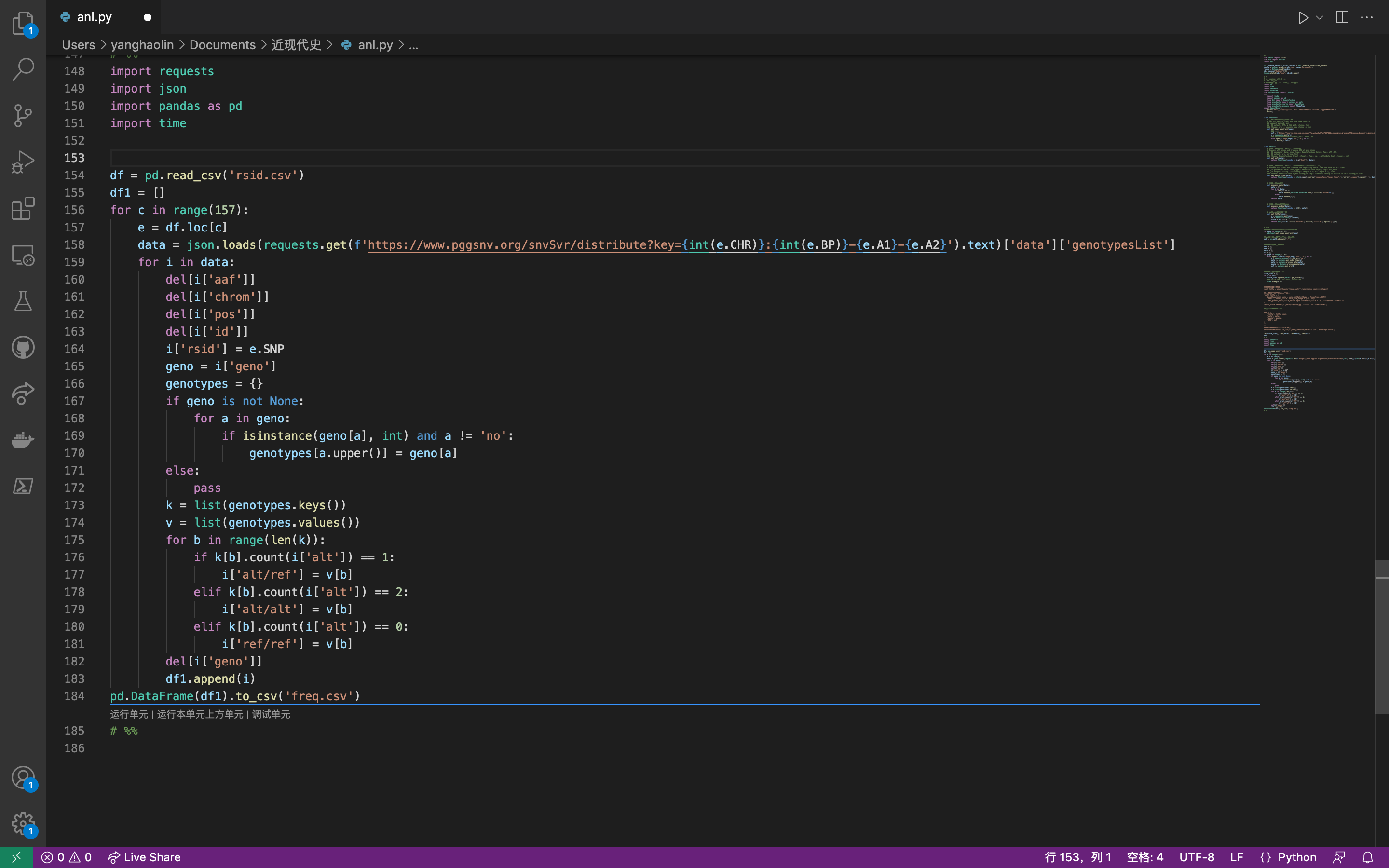
设计出的爬虫脚本如下:
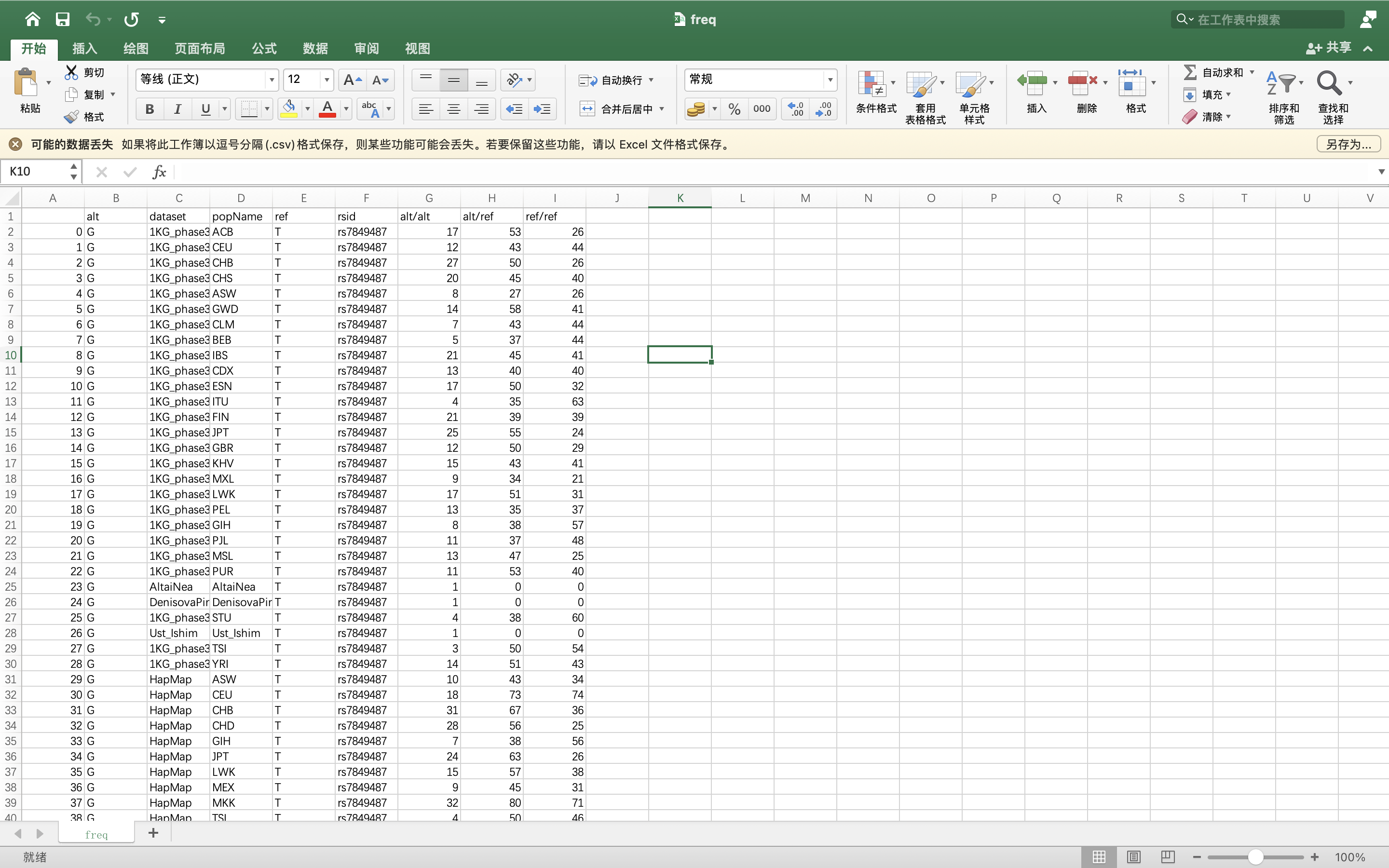
爬取效果如下:
reference:
【1】Demange, P.A., Malanchini, M., Mallard, T.T. et al. Investigating the genetic architecture of noncognitive skills using GWAS-by-subtraction. Nat Genet 53, 35–44 (2021). https://doi.org/10.1038/s41588-020-00754-2
【2】Chao Zhang#, Yang Gao#, Zhilin Ning#, Yan Lu#, Xiaoxi Zhang, Jiaojiao Liu, Bo Xie, Zhe Xue, Xiaoji Wang, Kai Yuan, Xueling Ge, Yuwen Pan, Chang Liu, Lei Tian, Yuchen Wang, Dongsheng Lu, Boon-Peng Hoh, Shuhua Xu*. PGG.SNV: understanding the evolutionary and medical implications of human single nucleotide variations in diverse populations. Genome Biology (2019), 20:215. https://doi.org/10.1186/s13059-019-1838-5
代码:
https://github.com/yhlhhhhh/HGG.SNV_Reptile
网站链接:https://www.pggsnv.org/
但是当我们要查询的数据量十分庞大时会比较费时间,所以我们写一段python脚本来批量获取数据。
首先使用chrome对该网站的基本情况进行分析。使用Chrome的开发者工具对传输数据进行监控,并将传输数据筛选为fetch和XHR模式,发现该网站的前后端是通过json传输的,属于前后端分离的情况,所以我们可以直接向网站拉去请求获取json。
我们将其中的json打开,发现里面确实含有位点相关信息,所以双击该json文件获取url。
url:https://www.pggsnv.org/snvSvr/distribute?key=1:231557623-G-C
接着我们对url进行解析。通过对比已知位点数据可知该url中的key应该是以这样的格式组成的:染色体号:碱基对位置-ref-alt
我们以《Nature-Genetics》期刊上的《Investigating the genetic architecture of noncognitive skills using GWAS-by-subtraction》【1】这篇文章的数据为例
设计出的爬虫脚本如下:
爬取效果如下:
reference:
【1】Demange, P.A., Malanchini, M., Mallard, T.T. et al. Investigating the genetic architecture of noncognitive skills using GWAS-by-subtraction. Nat Genet 53, 35–44 (2021). https://doi.org/10.1038/s41588-020-00754-2
【2】Chao Zhang#, Yang Gao#, Zhilin Ning#, Yan Lu#, Xiaoxi Zhang, Jiaojiao Liu, Bo Xie, Zhe Xue, Xiaoji Wang, Kai Yuan, Xueling Ge, Yuwen Pan, Chang Liu, Lei Tian, Yuchen Wang, Dongsheng Lu, Boon-Peng Hoh, Shuhua Xu*. PGG.SNV: understanding the evolutionary and medical implications of human single nucleotide variations in diverse populations. Genome Biology (2019), 20:215. https://doi.org/10.1186/s13059-019-1838-5
代码:
https://github.com/yhlhhhhh/HGG.SNV_Reptile


2 个回复
赞同来自:
赞同来自:
要回复问题请先登录或注册